O que é o sequenciamento de nova geração e para que serve?
Autores
Prabuddha Gupta, Shinjini Bandopadhyay, Soham Biswas, Subhrajit Barua
Jovens revisores

Resumo
Você sabia que o seu DNA pode ser lido? O DNA é um depósito de informações que faz de nós aquilo que somos. Mas, para realmente compreender essas informações, devemos primeiro aprender a lê-las. Os genes são partes do DNA que, em conjunto, funcionam como um manual de instruções que governa nossas células, assim como os códigos que dizem a um computador o que fazer. Por muitos anos, não havia nenhuma maneira para ler esses códigos genéticos. Na década de 1970, a invenção de uma tecnologia conhecida como sequenciamento de DNA tornou a leitura possível. O sequenciamento de DNA permitiu aos cientistas ler, compreender e comparar informações genéticas, o que constituiu um enorme avanço em nossa compreensão da biologia. Hoje, as tecnologias de sequenciamento desempenham um papel importante em quase todas as áreas, desde o tratamento de doenças até a agricultura. Na verdade, o sequenciamento de DNA foi crucial durante a pandemia da COVID-19, ajudando-nos a estudar o coronavírus e a desenvolver vacinas rapidamente.
A descoberta das bases ATGC
O ácido desoxirribonucleico (em inglês deoxyribonucleic acid,DNA), que é o material genético presente em todos os organismos vivos, foi descoberto em 1869 pelo pesquisador suíço Friedrich Miescher, que estava originalmente tentando estudar os glóbulos brancos. Em vez disso, ele isolou uma nova molécula, a que deu o nome de nucleína, em alusão aos núcleos das células. A estrutura da molécula de DNA foi identificada em 1953 por James Watson e Francis Crick, que contaram com algumas contribuições importantes de Rosalind Franklin [1].
A estrutura do DNA se assemelha a uma escada em caracol (Figura 1). Os corrimões laterais de cada degrau da escada são compostos por um grupo de fosfato (PO43-) e uma molécula de açúcar pentose (cinco carbonos), enquanto os degraus são formados por quatro substâncias chamadas bases nitrogenadas, a saber, adenina (A), guanina (G), citosina (C) e timina (T). Os emparelhamentos dessas bases são explicados pela regra de Chargaff, segundo a qual A sempre emparelha com T, e G sempre emparelha com C.
Juntos, uma molécula de fosfato, um açúcar pentose e uma base nitrogenada constituem o que é chamado de nucleotídeo. A disposição das bases no DNA é muito importante porque a sequência representa o código genético, do mesmo modo que a disposição adequada das letras do alfabeto forma palavras significativas. Essas “palavras” ou blocos de informação são os genes, que fornecem instruções para criar várias características, desde a cor dos olhos até o tipo de sangue. O termo coletivo para todos os genes presentes no corpo é genoma. Todos os membros de uma espécie têm genomas similares (mas não idênticos). O genoma humano apresenta propriedades que são exclusivas da espécie humana, mas pequenas variações nos genes tornam cada indivíduo um pouco diferente.

A primeira tentativa de decifrar o código: o sequenciamento de Sanger
Após o sucesso das descobertas de Watson e Crick, os cientistas tiveram uma compreensão clara da estrutura de uma molécula de DNA e de suas bases. Em seguida, surgiu a busca para determinar a ordem, ou a sequência, na qual as bases aparecem ao longo do comprimento do DNA. Esse processo é chamado de sequenciamento de DNA.
Em 1975, Frederick Sanger deu um grande passo no sequenciamento de DNA ao desenvolver um método chamado terminação de cadeia [2]. Nas nossas células, o DNA se replica para gerar cópias idênticas de si mesmo usando os nucleotídeos (A, T, G, C) como os blocos de construção. O método de Sanger baseava-se na apropriação do processo natural de replicação do DNA, adicionando nucleotídeos quimicamente alterados, cada um com um rótulo radioativo, além dos normais (depois, os rótulos radioativos foram substituídos por fluorescentes). Esses nucleotídeos modificados interrompiam a replicação do DNA em pontos aleatórios, sempre que eram incorporados à fita replicante do DNA. A replicação se encerrava de modo aleatório, criando vários fragmentos de DNA de diferentes comprimentos, cada um terminando com um nucleotídeo modificado, marcado radioativamente (ou codificado por cores).
Observando todos esses fragmentos, Sanger conseguiu descobrir a sequência da cadeia de DNA original, organizando-os na ordem correta, juntando os fragmentos do menor para o maior, a fim de visualizar a sequência correta do DNA. Por exemplo, o menor fragmento, com apenas um nucleotídeo, seria a versão modificada de A, T, G ou C. Isso lhe deixou claro que a primeira base do DNA original estava sendo sequenciada (Figura 2). O método de sequenciamento de Sanger abriu caminho para projetos de sequenciamento de DNA em grande escala, como o Projeto Genoma Humano.

O Projeto Genoma Humano
O Projeto Genoma Humano (PGH) foi um grande projeto internacional realizado por cientistas de todo o mundo, que iniciaram suas pesquisas em 1990 e trabalharam arduamente durante 13 longos anos para compreender o genoma humano. Eles completaram o sequenciamento do enorme genoma humano (contendo 3,2 bilhões de pares de bases) em 2003 – antes até do previsto! O PGH foi um evento marcante. Pela primeira vez, os cientistas puderam ler todo o modelo genético dos humanos, abrindo as portas para novas descobertas emocionantes. O sequenciamento do genoma humano completo foi um grande passo na melhoria dos tratamentos de saúde [3].
No entanto, é importante saber que as técnicas de sequenciamento de DNA utilizadas para alcançar esses resultados eram demoradas e ineficientes. Apesar dos resultados inovadores do PGH, o surgimento de tecnologias mais avançadas, como o sequenciamento de próxima geração (SNG), melhorou muito a precisão do sequenciamento de DNA, minimizando erros. O SNG revolucionou o setor de saúde ao reduzir o custo do sequenciamento e o tempo necessário para o diagnóstico. Hoje, todo o genoma humano pode ser sequenciado em apenas seis horas!
O que é o sequenciamento de nova geração?
Em 2006, uma empresa chamada Illumina anunciou um grande avanço no sequenciamento do DNA que poderia sequenciar simultaneamente um grande número de fitas de DNA. Essa alta velocidade permitiu que todo o genoma humano fosse sequenciado em menos de um dia. Esse método difere do método anterior de sequenciamento porque não depende da terminação da cadeia. Em vez disso, nesse novo método, sempre que uma nova base é adicionada à cadeia de DNA em replicação, é emitido um sinal fluorescente correspondente que é detectado em tempo real (Figura 3).
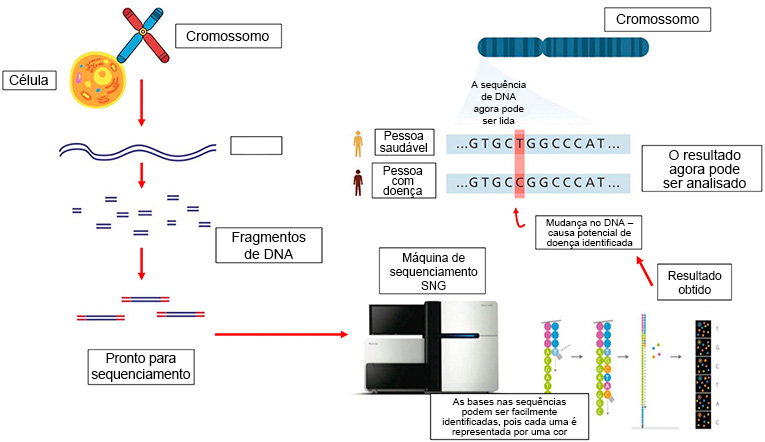
Como o sequenciamento de DNA é usado hoje?
Tratamento do câncer
O câncer sempre desafiou os médicos porque cada tipo de câncer é diferente, e um mesmo tipo pode atuar de formas distintas em pacientes diferentes. O câncer às vezes é causado por certos erros no DNA que podem ser identificados por sequenciamento. Os cientistas estão usando o SNG para analisar os genomas de pacientes, e para identificar os erros no DNA que são específicos de cada pessoa. Isso ajuda os médicos a prescrever tratamentos personalizados. Por exemplo, dois pacientes com o mesmo tipo de câncer podem responder de forma diferente ao mesmo medicamento – o tratamento que curou um falhará no outro. Pequenas alterações nos genomas de dois indivíduos podem ser detectadas usando-se o SNG e isso ajuda os médicos a entender como personalizar a terapia para indivíduos com base em suas diferenças genéticas [4]. Esse tipo de tratamento personalizado é chamado de medicina personalizada.
Evolução
A vida na Terra começou como organismos unicelulares, que foram se tornando cada vez mais complexos. Os animais são as formas de vida mais complexas da Terra. O SNG tem sido usado para comparar o genoma humano com o genoma de outros animais e isso pode nos dizer, em detalhes fascinantes, até que ponto estamos relacionados a eles. Além disso, mutações pequenas (alterações no DNA) que existem nos organismos da mesma espécie são uma parte importante da evolução porque cada mutação é capaz de alterar permanentemente uma parte do genoma dessa espécie. O SNG permite aos cientistas estudarem como as mutações se acumularam ao longo da evolução.
Vacinas
Desde a investigação da origem do vírus SARS-CoV2 até o rápido desenvolvimento das vacinas, o sequenciamento tem sido uma ajuda tremenda durante a pandemia da COVID-19. As vacinas ensinam o sistema imunológico a reconhecer ameaças e erradicá-las. O SNG ajudou os cientistas a sequenciarem o SARS-CoV2, levando-os a aprender mais sobre as características genéticas do vírus que ameaça o sistema imunológico, como a proteína spike. O SNG também foi útil no rastreamento de novas variantes virais – os vírus também evoluem!
Alergias
Você provavelmente já ouviu falar sobre alergia ao glúten ou intolerância à lactose. O SNG ajuda os cientistas a identificar se um indivíduo tem a probabilidade de ser alérgico ao glúten ou intolerante à lactose, analisando seu genoma. Certas sequências de DNA podem aumentar a probabilidade de uma pessoa desenvolver alergias.
Micróbios úteis
Nem todos os micróbios que vivem dentro do corpo são prejudiciais. Bons micróbios nos ajudam a combater infecções. A microbiota intestinal é uma comunidade de micróbios que vive no trato digestivo humano e pode ser considerada nosso
“segundo genoma” porque influencia o sistema imunológico e a saúde geral [5]. A microbiota intestinal não é fácil de estudar em laboratórios, mas o SNG pode ser usado para analisar os genomas desses organismos e entender as suas funções. O American Gut Project1 foi a maior iniciativa de ciência cidadã do mundo a analisar os micróbios que viviam nas entranhas de cerca de 11.000 pessoas. E foi possível graças ao SNG.
Conclusão
Em resumo, a sequenciação do DNA revolucionou a pesquisa em ciências da vida e abriu caminho para cuidados de saúde mais precisos e eficientes. O SNG tornou essa tarefa mais rápida e acessível. Nos últimos anos, foram desenvolvidas técnicas mais avançadas de sequenciamento de DNA, como o Oxford Nanopore. Esses métodos são até mais rápidos e mais precisos! No entanto, embora possamos sequenciar o DNA, ainda não foi inventado nenhum método para sequenciar proteínas. Esse é um dos desenvolvimentos mais esperados da biologia. Quem sabe um de vocês, jovens leitores, consiga realizar essa grande façanha!
Glossário
Bases nitrogenadas: Moléculas que contêm nitrogênio e são componentes importantes do DNA.
Nucleotídeo: Componente do DNA que contém uma molécula de fosfato, uma molécula de açúcar e uma base nitrogenada.
Gene: Parte da sequência do DNA que contêm o código responsável pela criação de uma proteína.
Genoma: Conjunto completo de genes em um organismo vivo.
Sequenciamento de DNA: Processo de determinação da sequência de bases nitrogenadas em um fragmento de DNA.
Projeto Genoma Humano: Esforço de pesquisa internacional que visava a determinar a sequência de todos os genes do corpo humano.
Sequenciamento de próxima geração: Moderno e rápido sequenciamento de DNA que pode ler um grande volume de sequências de DNA simultaneamente.
Mutação: Qualquer mudança ou alteração na sequência de DNA que pode ou não mudar a função do gene.
Conflito de interesses
Os autores declaram que a pesquisa foi realizada sem nenhuma relação financeira ou comercial capaz de gerar um conflito de interesses.
Referências
[1] Watson, J. e Crick, F. 1953. “Molecular structure of nucleic acids: a structure for deoxyribose nucleic acid.” Nature 171:737–38. DOI: 10.1038/171737a0.
[2] Heather, J. M. e Chain, B.2016. “The sequence of sequencers: the history of sequencing DNA.” Genomics 107:1–8. DOI: 10.1016/j.ygeno.2015.11.003.
[3] Green, E. D., Watson, J. D. e Collins, F. S. 2015. “Human Genome Project: twenty-five years of big biology.” Nature 526:29–31. DOI: 10.1038/526029a.
[4] Gupta, A. K. e Gupta, U. D. 2014. “Next generation sequencing and its applications”, em Animal Biotechnology (Elsevier), pp. 345–67. DOI: 10.1016/b978-0-12-416002-6.00019-5.
[5] Cao, Y., Fanning, S., Pross, S., Jordan, K. e Srikumar, S. 2017. “A review on the applications of next-generation sequencing technologies as applied to food-related microbiome studies.” Front. Microbiol. 8:1829. DOI: 10.3389/fmicb.2017.01829.
Citação
Barua, S., Bandopadhyay, S., Biswas, S. e Gupta, P. (2022). “What is next-generation sequencing and why do we need it?”. Front. Young Minds. 10:746502. DOI: 10.3389/frym.2022.746502.
Este é um artigo de acesso aberto distribuído sob os termos da Creative Commons Attribution License (CC BY). O uso, distribuição ou reprodução em outros fóruns é permitido, desde que o(s) autor(es) original(is) e o(s) proprietário(s) dos direitos autorais sejam creditados e que a publicação original nesta revista seja citada, de acordo com a prática acadêmica aceita. Não é permitido nenhum uso, distribuição ou reprodução que não esteja em conformidade com estes termos.
Encontrou alguma informação errada neste texto?
Entre em contato conosco pelo e-mail:
parajovens@unesp.br


